细胞STR鉴定:科研中的“基因身份证”
一串微卫星DNA的重复次数,决定了你的细胞是否还是“它自己”。
在生命科学研究中,一个触目惊心的数据常被忽略:全球超过600种细胞系被确认存在命名错误或交叉污染,其中约30%的细胞实验因污染或身份错误导致数据失真,甚至引发论文撤稿。2015年,因误用被HeLa细胞污染的细胞系,一位科学家被迫撤销了发表在《Nature》的论文。细胞身份危机不仅造成数十亿美元的经济损失,更侵蚀着科研的根基。近年来,美国国立卫生研究院(NIH)、美国典型培养物保藏中心(ATCC)、《Nature》和《Science》等权威机构和顶级学术期刊纷纷发出呼吁,要求研究者对细胞进行严格的鉴定。目前,越来越多的学术期刊在投稿时明确要求提供细胞的STR分型图谱。
那么,什么是STR呢?
STR(短串联重复序列)是一类广泛存在于真核生物基因组中的特殊DNA序列。由长度为3~7个碱基对的短串连重复序列组成,不同个体在同一STR位点的重复次数存在显著差异。这些重复序列广泛存在于人类基因组中, 可作为高度多态性标记。
STR鉴定一般步骤
STR鉴定的本质是通过PCR(聚合酶链式反应)扩增检测特定位点的重复拷贝数,构建细胞的基因身份图谱。其流程标准化程度高,需经历五个关键步骤:
1)DNA提取
取≥10⁶个细胞(活细胞/冻存细胞/细胞沉淀),提取高纯度DNA(OD260/280=1.8~2.0)
2)STR位点选择
常用人源细胞STR检测包含21个核心位点,大多数检测平台会选用10~21个位点进行联合检测,以下是典型人类STR位点:
STR位点 | 染色体定位 | 功能 |
TH01 | 11p15.5 | 高多态性 |
D5S818 | 5q23.2 | 稳定性强 |
AMEL | X/Y | 性别鉴定 |
vWA | 12p13.31 | 经典STR位点 |
TPOX | 2p25 | 常用鉴定位点 |
3)PCR扩增
使用多重荧光PCR技术(FAM、HEX、TAMRA等荧光通道)扩增特定长度的STR序列
4)毛细管电泳
采用ABI 3500/3730xl等系统通过分子大小分离STR片段,激光激发荧光信号,生成电泳峰图
5)数据分析
软件(GeneMapper)读取峰值,对应等位基因编号,与ATCC、DSMZ、Cellosaurus数据库比对,生成完整STR图谱与相似度报告
如何进行STR报告解读
一般检测出具报告如下:

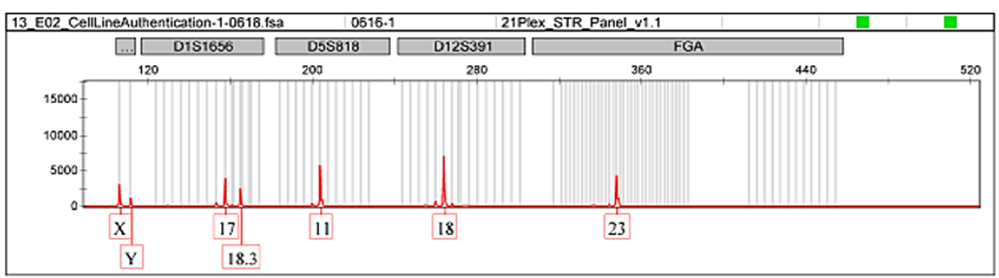
该报告展示了EnkiLife的A549细胞株(货号CXH00001)人源细胞系的STR鉴定的部分结果,摘取报告解读结果如下:
STR位点 | 等位基因 | 峰数 | 类型 |
D3S1358 | 16/16 | 1 | 纯合 |
vWA | 14/14 | 1 | 纯合 |
D7S820 | 8/11 | 2 | 杂合 |
CSF1PO | 10/12 | 2 | 杂合 |
AMEL | X/Y | 2 | 男性 |
提交的细胞STR分型图谱在DSMZ数据库中与人类细胞系在9个位点上完全匹配:A549
1. 异常信号预警:
三峰现象:≥3个峰 → 提示细胞交叉污染
AMEL-Y缺失:仅106bp峰 → 女性细胞源
多峰化:多细胞混杂
2. 相似度匹配
理论上一个基因位点只会出现1个或2个峰,即纯合和杂合
与数据库匹配,相似度>90% , 判定为同一细胞系
与数据库匹配,相似度80-90%之间,待测细胞系与标准细胞系具有相关性,建议复核
与数据库匹配,相似度在50%-80%之间,需验证来源,结合细胞系形态、细胞系特异性标记物等做辅助分析并进行综合判断
与数据库匹配,相似度<50% ,与对应细胞株不相关,弃用并溯源
注:小鼠细胞因近交系特性,匹配度80–85%仍可能同源,需结合遗传背景研判
当前,STR技术存在着明显边界,如STR仅适用于人源和小鼠细胞的鉴定;存在着数据库盲区,未收录STR数据的原代细胞只能做排他性鉴定;近交系误判,小鼠近交系相似度阈值需下调等问题。但是随着NIH、ATCC及顶级期刊的联合推动,STR鉴定已成为细胞实验不可绕过的质量闸门。2025年全球STR鉴定市场预计达数亿美元规模,中国增速领跑全球。
这项技术背后承载的不仅是实验数据的可靠性,更是科学精神的底线——让每一株细胞都能被追溯,每一个结果都经得起重复验证。正如国际细胞系认证委员会(ICLAC)所呼吁:“没有STR认证的细胞实验,如同没有地基的摩天大楼‘’
参考文献
1. Reid, Y.A., Characterization and authentication of cancer cell lines: an overview. Methods. Mol Biol, 2011. 731: p. 35-43.
2. Capes-Davis, A., et al., Check your cultures! A list of cross-contaminated or misidentified cell lines. Int J Cancer, 2010. 127(1): p. 1-8.
3. Chatterjee, R., Cell biology. Cases of mistaken identity. Science, 2007. 315(5814): p. 928-31.
4. Neimark, J., Line of attack. Science, 2015. 347(6225): p. 938-40.
5. Masters, J.R., et al., Short tandem repeat profiling provides an international reference standard for human cell lines. Proc Natl Acad Sci U S A, 2001. 98(14): p. 8012-7.
6. American Type Culture Collection Standards Development Organization Workgroup, A.S.N., Cell line misidentification: the beginning of the end. Nat Rev Cancer, 2010. 10(6): p. 441-8.
7. American Type Culture Collection Standards Development Organization Workgroup,A.S.N., Authentication of Human Cell Lines: Standardization of STR Profiling. 2011,ANSI/ATCC ASN-0002-2011.
8. Edwards, A., et al., DNA typing and genetic mapping with trimeric and tetrameric tandem repeats. Am J Hum Genet, 1991. 49(4): p. 746-56.
 | Dylan Z Dylan Z是EnkiLife的蛋白抗体研发专家,精通蛋白表达系统和抗体制备技术,在技术上精益求精,致力于为用户开发稳定好用的产品。 |
